Contenido
- Información genética en procariotas
- Etapas en la expresión génica
- Transcripción en células bacterianas
- Transcripción: Fase de Iniciación
- Transcripción: Fase de alargamiento
- Transcripción: Fase de Terminación
- Traducción en células bacterianas
- Traducción: Iniciación
- Traducción: alargamiento
- Traducción: Terminación
- Traducción y antibióticos
- Procesamiento de proteínas posterior a la traducción
- Fosforilación de proteínas
- Acetilación de proteínas y glicosilación
- Expresión génica en Archaea
Los procariotas son organismos vivos pequeños y unicelulares. Son uno de los dos tipos de células comunes: procariota y eucariota.
Dado que las células procariotas no tienen núcleo u orgánulos, la expresión génica se produce en el citoplasma abierto y todas las etapas pueden ocurrir simultáneamente. Aunque los procariotas son más simples que los eucariotas, controlar la expresión génica sigue siendo crucial para su comportamiento celular.
Información genética en procariotas
Los dos dominios de los procariotas son las bacterias y las arqueas. Ambos carecen de un núcleo definido, pero aún tienen un código genético y ácidos nucleicos. Aunque no hay cromosomas complejos como los que vería en las células eucariotas, los procariotas tienen piezas circulares de ácido desoxirribonucleico (ADN) ubicado en el nucleoide.
Sin embargo, no hay membrana alrededor del material genético. En general, los procariotas tienen menos secuencias no codificantes en su ADN en comparación con los eucariotas. Esto puede deberse a que las células procariotas son más pequeñas y tienen menos espacio para una molécula de ADN.
los nucleoide es simplemente la región donde vive el ADN en la célula procariota. Tiene una forma irregular y puede variar en tamaño. Además, el nucleoide está unido a la membrana celular.
Los procariotas también pueden tener ADN circular llamado plásmidos. Es posible que tengan uno o más plásmidos en una célula. Durante la división celular, los procariotas pueden pasar por la síntesis de ADN y la separación de plásmidos.
En comparación con los cromosomas en eucariotas, los plásmidos tienden a ser más pequeños y tienen menos ADN. Además, los plásmidos pueden replicarse por sí mismos sin otro ADN celular. Algunos plásmidos llevan los códigos de genes no esenciales, como los que le dan a las bacterias su resistencia a los antibióticos.
En ciertos casos, los plásmidos también pueden moverse de una célula a otra y compartir información como la resistencia a los antibióticos.
Etapas en la expresión génica
La expresión génica es el proceso a través del cual la célula traduce el código genético en aminoácidos para la producción de proteínas. A diferencia de los eucariotas, las dos etapas principales, que son la transcripción y la traducción, pueden ocurrir al mismo tiempo en los procariotas.
Durante la transcripción, la célula traduce el ADN en una molécula de ARN mensajero (ARNm). Durante la traducción, la célula produce los aminoácidos del ARNm. Los aminoácidos formarán las proteínas.
Tanto la transcripción como la traducción ocurren en los procariotas. citoplasma. Al hacer que ambos procesos sucedan al mismo tiempo, la célula puede producir una gran cantidad de proteína a partir de la misma plantilla de ADN. Si la célula ya no necesita la proteína, entonces la transcripción puede detenerse.
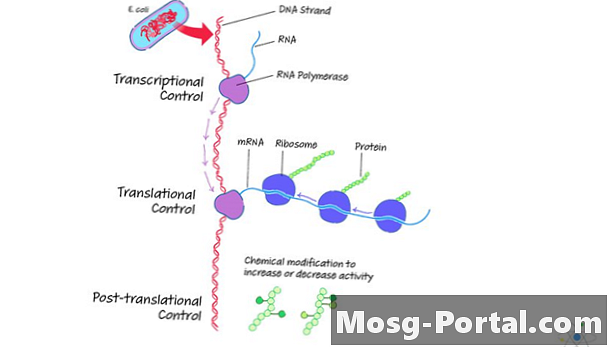
Transcripción en células bacterianas
El objetivo de la transcripción es crear una cadena complementaria de ácido ribonucleico (ARN) a partir de una plantilla de ADN. El proceso tiene tres partes: iniciación, alargamiento y terminación de la cadena.
Para que ocurra la fase de iniciación, el ADN tiene que desenrollarse primero y el área donde esto sucede es el burbuja de transcripción.
En las bacterias, encontrará la misma ARN polimerasa responsable de toda la transcripción. Esta enzima tiene cuatro subunidades. A diferencia de los eucariotas, los procariotas no tienen factores de transcripción.
Transcripción: Fase de Iniciación
La transcripción comienza cuando el ADN se desenrolla y la ARN polimerasa se une a un promotor. Un promotor es una secuencia especial de ADN que existe al comienzo de un gen específico.
En bacterias, el promotor tiene dos secuencias: -10 y -35 elementos. El elemento -10 es donde generalmente se desenrolla el ADN, y se encuentra a 10 nucleótidos del sitio de iniciación. El elemento -35 está a 35 nucleótidos del sitio.
La ARN polimerasa se basa en una cadena de ADN para ser la plantilla, ya que construye una nueva cadena de ARN llamada transcripción de ARN. La cadena de ARN resultante o la transcripción primaria es casi la misma que la cadena de ADN no moldeadora o codificante. La única diferencia es que todas las bases de timina (T) son bases de uracilo (U) en el ARN.
Transcripción: Fase de alargamiento
Durante la fase de elongación de la cadena de la transcripción, la ARN polimerasa se mueve a lo largo de la cadena de la plantilla de ADN y forma una molécula de ARNm. La cadena de ARN se alarga a medida que se agregan más nucleótidos.
Esencialmente, la ARN polimerasa camina a lo largo del soporte de ADN en la dirección 3 a 5 para lograr esto. Es importante tener en cuenta que las bacterias pueden crear ARNm policistrónicos ese código para múltiples proteínas.
••• SciencingTranscripción: Fase de Terminación
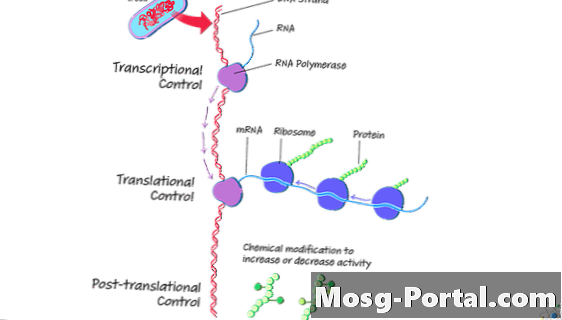
Durante la fase de terminación de la transcripción, el proceso se detiene. Hay dos tipos de fases de terminación en procariotas: terminación dependiente de Rho y terminación independiente de Rho.
En Terminación dependiente de Rho, un factor de proteína especial llamado Rho interrumpe la transcripción y la termina. El factor de proteína Rho se une a la cadena de ARN en un sitio de unión específico. Luego, se mueve a lo largo de la cadena para alcanzar la ARN polimerasa en la burbuja de transcripción.
A continuación, Rho separa la nueva cadena de ARN y la plantilla de ADN, por lo que finaliza la transcripción. La ARN polimerasa deja de moverse porque alcanza una secuencia de codificación que es el punto de parada de la transcripción.
En Terminación independiente de Rho, la molécula de ARN forma un bucle y se desprende. La ARN polimerasa alcanza una secuencia de ADN en el filamento molde que es el terminador y tiene muchos nucleótidos de citosina (C) y guanina (G). La nueva cadena de ARN comienza a plegarse en forma de horquilla. Sus nucleótidos C y G se unen. Este proceso detiene el movimiento de la ARN polimerasa.
Traducción en células bacterianas
La traducción crea una molécula de proteína o polipéptido basado en la plantilla de ARN creada durante la transcripción. En las bacterias, la traducción puede ocurrir de inmediato y, a veces, comienza durante la transcripción. Esto es posible porque los procariotas no tienen membranas nucleares ni orgánulos para separar los procesos.
En eucariotas, las cosas son diferentes porque la transcripción ocurre en el núcleo y la traducción está en el citosol, o líquido intracelular, de la célula. Un eucariota también usa ARNm maduro, que se procesa antes de la traducción.
Otra razón por la cual la traducción y la transcripción pueden ocurrir al mismo tiempo en bacterias es que el ARN no necesita el procesamiento especial que se ve en los eucariotas. El ARN bacteriano está listo para la traducción inmediata.
La cadena de ARNm tiene grupos de nucleótidos llamados codones. Cada codón tiene tres nucleótidos y códigos para una secuencia de aminoácidos específica. Aunque solo hay 20 aminoácidos, las células tienen 61 codones para aminoácidos y tres codones de parada. AUG es el codón de inicio y comienza la traducción. También codifica el aminoácido metionina.
Traducción: Iniciación
Durante la traducción, la cadena de ARNm actúa como una plantilla para producir aminoácidos que se convierten en proteínas. La célula decodifica el ARNm para lograr esto.
La iniciación requiere transferencia de ARN (ARNt), un ribosoma y ARNm. Cada molécula de ARNt tiene un anticodón para un aminoácido El anticodón es complementario al codón. En las bacterias, el proceso comienza cuando una pequeña unidad ribosómica se une al ARNm en un Secuencia Shine-Dalgarno.
La secuencia Shine-Dalgarno es un área de unión ribosómica especial tanto en bacterias como en arqueas. Usualmente lo ves alrededor de ocho nucleótidos desde el codón de inicio AUG.
Como los genes bacterianos pueden tener transcripción en grupos, un ARNm puede codificar muchos genes. La secuencia Shine-Dalgarno hace que sea más fácil encontrar el codón de inicio.
Traducción: alargamiento
Durante el alargamiento, la cadena de aminoácidos se alarga. Los ARNt añaden aminoácidos para formar la cadena polipeptídica. Un tRNA comienza a trabajar en el Sitio P, que es una parte media del ribosoma.
Al lado del sitio P está el Un sitio. Un ARNt que coincide con el codón puede ir al sitio A. Entonces, se puede formar un enlace peptídico entre los aminoácidos. El ribosoma se mueve a lo largo del ARNm, y los aminoácidos forman una cadena.
Traducción: Terminación
La terminación ocurre debido a un codón de parada. Cuando un codón de parada ingresa al sitio A, el proceso de traducción se detiene porque el codón de parada no tiene un ARNt complementario. Proteínas llamadas factores de liberación que encajan en el sitio P pueden reconocer los codones de parada y evitar que se formen enlaces peptídicos.
Esto sucede porque los factores de liberación pueden hacer que las enzimas agreguen una molécula de agua, lo que hace que la cadena se separe del ARNt.
Traducción y antibióticos
Cuando toma algunos antibióticos para tratar una infección, pueden funcionar al interrumpir el proceso de traducción en bacterias. El objetivo de los antibióticos es matar las bacterias y evitar que se reproduzcan.
Una forma de lograr esto es afectar los ribosomas en las células bacterianas. Los medicamentos pueden interferir con la traducción del ARNm o bloquear la capacidad de la célula para formar enlaces peptídicos. Los antibióticos pueden unirse a los ribosomas.
Por ejemplo, un tipo de antibiótico llamado tetraciclina puede ingresar a la célula bacteriana al cruzar la membrana plasmática y acumularse dentro del citoplasma. Luego, el antibiótico puede unirse a un ribosoma y bloquear la traducción.
Otro antibiótico llamado ciprofloxacina afecta la célula bacteriana al atacar una enzima responsable de desenrollar el ADN para permitir la replicación. En ambos casos, las células humanas se salvan, lo que permite a las personas usar antibióticos sin matar sus propias células.
Tema relacionado: organismos multicelulares
Procesamiento de proteínas posterior a la traducción
Una vez finalizada la traducción, algunas células continúan procesando las proteínas. Modificaciones postraduccionales (PTM) de proteínas permiten a las bacterias adaptarse a su entorno y controlar el comportamiento celular.
En general, las PTM son menos comunes en procariotas que en eucariotas, pero algunos organismos las tienen. Las bacterias también pueden modificar las proteínas y revertir los procesos. Esto les da más versatilidad y les permite utilizar la modificación de proteínas para la regulación.
Fosforilación de proteínas
Fosforilación de proteínas Es una modificación común en las bacterias. Este proceso implica agregar un grupo fosfato a la proteína, que tiene átomos de fósforo y oxígeno. La fosforilación es esencial para la función proteica.
Sin embargo, la fosforilación puede ser temporal porque es reversible. Algunas bacterias pueden usar la fosforilación como parte del proceso para infectar a otros organismos.
La fosforilación que ocurre en las cadenas laterales de aminoácidos de serina, treonina y tirosina se llama Ser / Thr / Tyr fosforilación.
Acetilación de proteínas y glicosilación
Además de las proteínas fosforiladas, las bacterias pueden tener acetilado y glicosilado proteínas También pueden tener metilación, carboxilación y otras modificaciones. Estas modificaciones juegan un papel importante en la señalización celular, la regulación y otros procesos en bacterias.
Por ejemplo, la fosforilación de Ser / Thr / Tyr ayuda a las bacterias a responder a los cambios en su entorno y a aumentar las posibilidades de supervivencia.
La investigación muestra que los cambios metabólicos en la célula están asociados con la fosforilación de Ser / Thr / Tyr, lo que indica que las bacterias pueden responder a su entorno cambiando sus procesos celulares. Además, las modificaciones postraduccionales les ayudan a reaccionar de manera rápida y eficiente. La capacidad de revertir cualquier cambio también proporciona un control significativo.
Expresión génica en Archaea
Las arqueas usan mecanismos de expresión génica que son más similares a los eucariotas. Aunque las arqueas son procariotas, tienen algunas cosas en común con las eucariotas, como la expresión génica y la regulación génica. Los procesos de transcripción y traducción en arqueas también tienen algunas similitudes con las bacterias.
Por ejemplo, tanto las arqueas como las bacterias tienen metionina como primer aminoácido y AUG como codón de inicio. Por otro lado, tanto las arqueas como los eucariotas tienen un Caja TATA, que es una secuencia de ADN en el área del promotor que muestra dónde decodificar el ADN.
La traducción en arqueas se asemeja al proceso visto en bacterias. Ambos tipos de organismos tienen ribosomas que consisten en dos unidades: las subunidades 30S y 50S. Además, ambos tienen ARNm policistrónicos y secuencias de Shine-Dalgarno.
Existen múltiples similitudes y diferencias entre bacterias, arqueas y eucariotas. Sin embargo, todos dependen de la expresión génica y la regulación génica para sobrevivir.